Cre(Cyclization Recombination Enzyme)是一種重組酶,來源于P1噬菌體,其基因編碼區序列全長1029bp,為38kDa大小的、由343個氨基酸組成的多肽單體蛋白。Cre重組酶的C-末端結構域包含催化活性位點,能夠催化DNA分子中特定位點之間的重組,同時,Cre還能識別特異的DNA序列,即loxP位點,使兩個loxP位點間發生基因重組。
LoxP是Locus of X-overP1的縮寫,是位于P1噬菌體中長度為34bp的一段序列,由兩個13bp的反向回文序列和8bp的中間間隔序列共同組成,反向回文序列是Cre重組酶的識別和結合區域,間隔序列決定LoxP序列的方向。
Cre/loxP誘導基因重組的方式
一般而言,當細胞基因組內存在兩個LoxP位點時,Cre重組酶會誘導兩個LoxP位點間的序列發生重組。重組的結果取決于兩個loxP位點的方向,主要有以下幾種可能:
① 如果兩個LoxP位點位于一條DNA鏈上且方向相同,Cre重組酶能有效地刪除兩個LoxP位點間的序列(Deletion)(圖1A);
② 如果兩個LoxP位點位于一條DNA鏈上但方向相反,Cre重組酶能誘導兩個LoxP位點間的序列翻轉(Inversion)(圖1B);
③如果兩個LoxP位點分別位于兩條不同的DNA鏈或染色體上,Cre重組酶能誘導兩條DNA鏈發生交換或染色體易位,即基因轉座(Translocation)(圖1C)
④如果四個loxP位點分別位于兩條不同的DNA鏈或染色體上,Cre重組酶能誘導loxP間的序列互換(cassette exchange)(圖1D)。
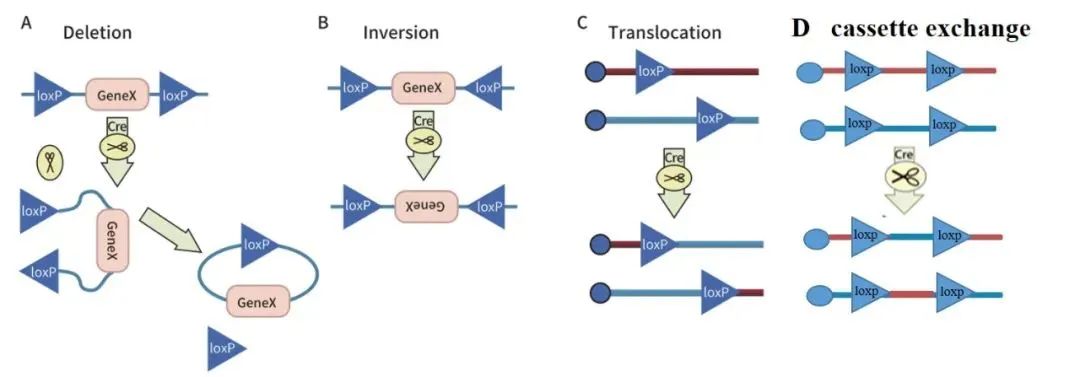
圖1 Cre-loxP誘導基因重組的方式
Cre-LoxP系統可以實現對基因的操控,根據Cre重組系統誘導基因重組的方式,我們可以通過如下三種策略實現Cre依賴的基因表達:
1、LSL序列(條件性基因選擇)
將LoxP2和轉錄終止信號盒(Transcription STOP cassette)插入啟動子和目的基因之間,轉錄終止信號盒兩端各有一個同向的LoxP位點,組成LoxP-STOP-LoxP-gene模式,即LSL序列。
此情況下,在無Cre酶存在的細胞中,轉錄終止信號盒下游靶基因完全不表達,但如果細胞中含有Cre酶,基因重組中的Deletion過程發生,移除轉錄終止信號盒,進而表達目的基因。這是一種簡單有效的辦法,我們可以通過LSL的設置,有選擇性地在某些細胞中表達基因。
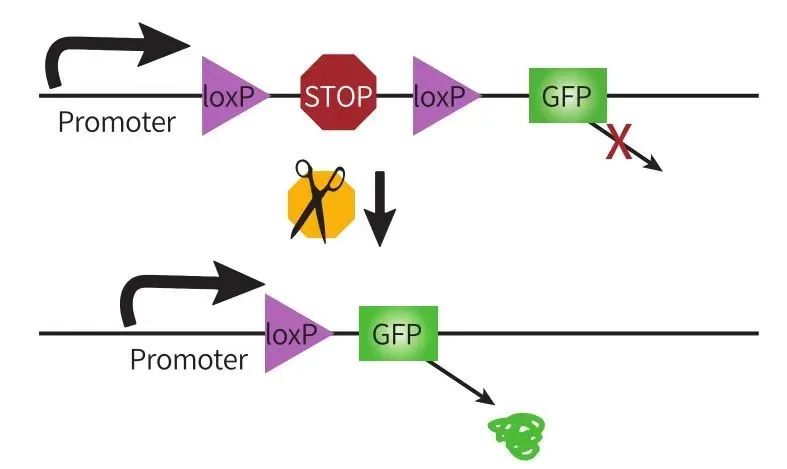
圖2 Cre依賴的基因表達-LSL策略
2、DIO/DO序列
通過引入兩對不相容的反向Lox位點LoxP和Lox2272,經過兩組Lox位點的兩輪重組可達到一種穩定狀態。也就是可以通過Cre重組酶的存在與否來控制基因的表達。
這種策略被稱為DIO(Cre-on)或DO(Cre-off)。在這種策略下,任一對Lox位點間的序列會在Cre的作用下發生可逆的快速翻轉,之后Cre重組酶馬上不可逆地切割翻轉后的同向Lox位點,只留下翻轉后的基因和單獨的LoxP、Lox2272位點,防止基因的再次翻轉。這種巧妙的設計讓科研人員可以在病毒載體中構建DIO和反向基因,感染Cre陽性細胞后,可以讓Cre陽性的細胞表達基因,是為DIO結構;如果預先包裝的基因是正向,那么Cre陽性的細胞則不表達基因,其余細胞表達基因,是為DO結構。因此,人為操控基因表達變成了現實。
?
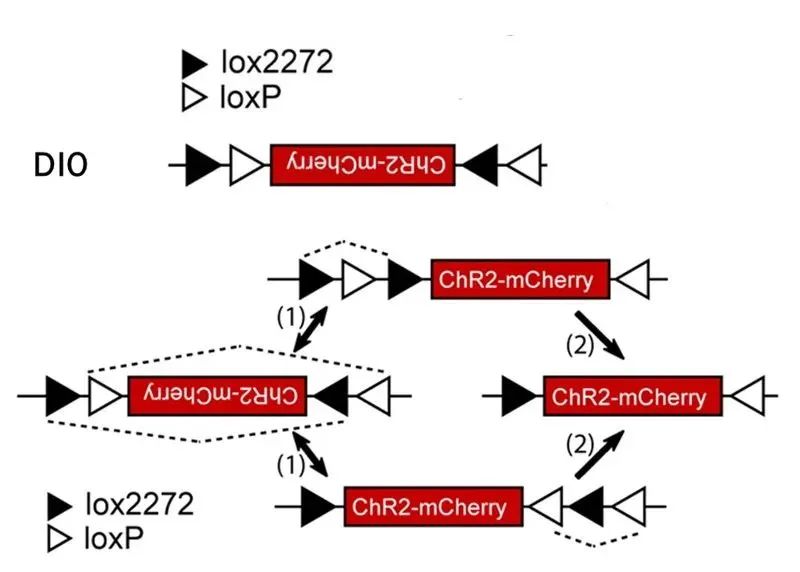
圖3借助LoxP和Lox2272的FLEX策略實現Cre依賴的基因表達
(Scott M. Sternson, et al., J. Neurosci., 2008)
3、MADM系統
雙標記嵌合體分析MADM(mosaic analysis with the double markers)系統,是基于Cre誘導發生Translocation過程實現的。通過同源重組將兩個相互嵌合的標記基因(熒光蛋白)分別定位到同源染色體上的相同位點,位于同一條DNA鏈上的熒光蛋白N端(C端)和另一種熒光蛋白N端(C端)之間插入一個Loxp位點。在沒有Cre的情況下,不表達功能性的GFP或RFP;但在特定時期、特定細胞中表達Cre時,Cre能誘導帶有兩種不同熒光蛋白N端的DNA鏈與另一條帶有相同熒光蛋白C端的DNA鏈在細胞有絲分裂G2期發生重組,使得兩個子代細胞分別表達有任一種有功能性的熒光蛋白(稱為:X-segregation),或者其中一個子代細胞表達兩種熒光蛋白而另一種子代細胞不帶任何功能性熒光蛋白(稱為:Z-segregation)。另外,重組也可能發生在細胞周期的G1期或者G0期,在這種情況下,細胞同時表達兩種熒光蛋白。
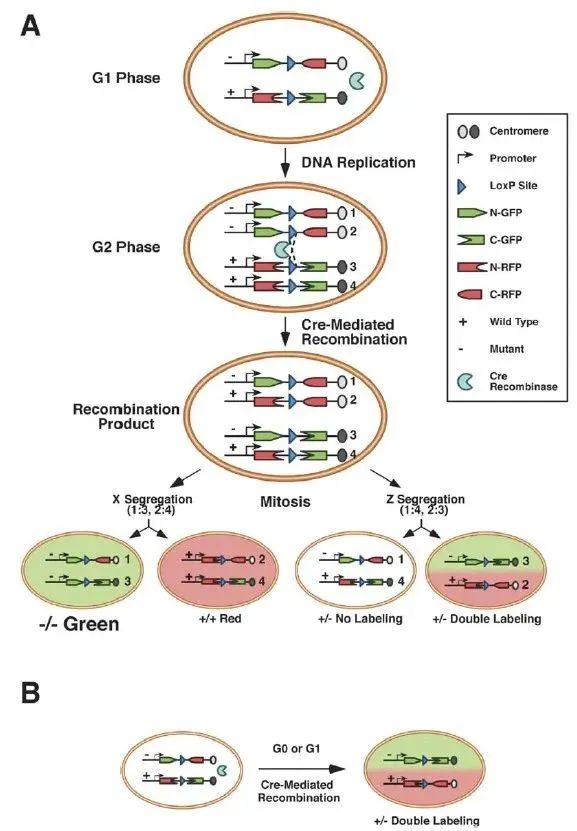
圖4 MADM系統
(Zong Hui, et al., Cell., 2005)
Cre/loxp系統的應用
通過病毒引入Cre或loxP元件,結合一種轉基因小鼠可達到對某類細胞進行特異性標記或基因操作的目的。
LSL策略應用
mT/mG(ROSA-pCA-loxp-mT-pA-loxp-mG-pA)鼠是一種雙熒光報告鼠,這種鼠在正常情況下表達定位在膜上的紅色熒光蛋白(TdTomato),而當細胞表達Cre后,則表達定位在膜上的綠色熒光蛋白(GFP)。
Alb為成年動物肝細胞特異性啟動子,將rAAV2/8-Alb-Cre病毒尾靜脈注射mT/mG小鼠,可發現特異性感染小鼠肝臟組織(綠色),而心臟組織未被感染(表現為紅色)。
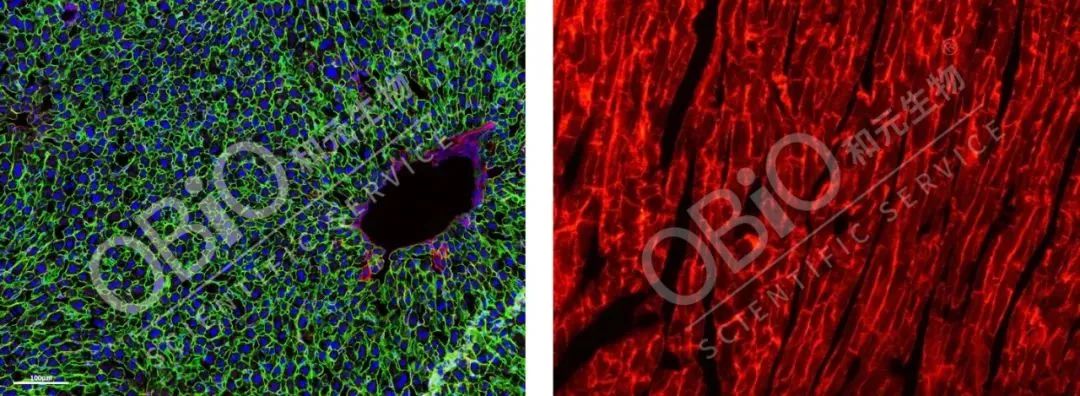
圖5肝臟組織(左)、心臟組織(右)
?
|
病毒產品 |
rAAV2/8-Alb-Cre |
|
實驗動物 |
mT/mG鼠 |
|
注射方式 |
尾靜脈注射 |
|
感染部位 |
肝臟組織 |
|
病毒用量 |
2E+11VG/只 |
?
管吉松教授團隊將AAV-hSyn-Cre注射于Ash1lfl/fl小鼠右側AUD區條件性敲除Ash1l基因,發現Ash1l以細胞自主效應的方式介導皮層神經元的活動依賴的突觸修剪過程。(點擊閱讀:Neuron | 拯救星星之子!上科大管吉松組揭示ASH1L單倍不足致小鼠自閉癥的神經機制)
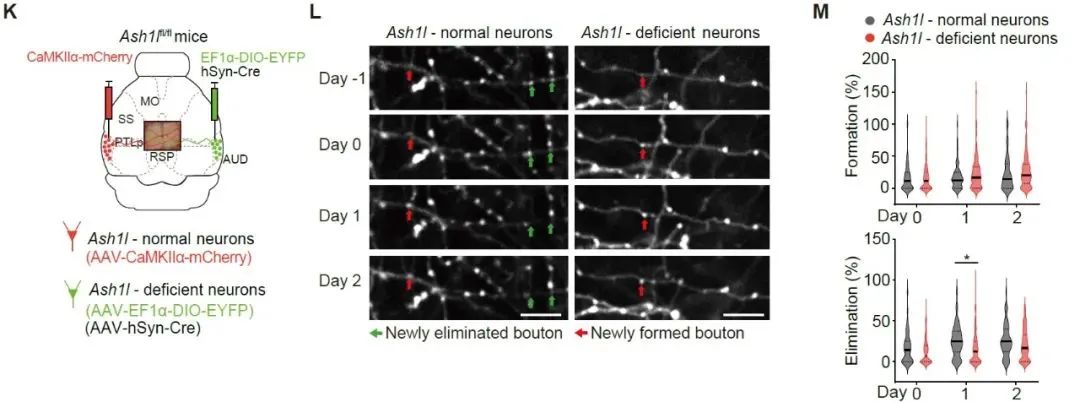
圖6 AAV-Cre注射Flox小鼠實現條件性敲除
?(Yan Yuze, et al., Neuron., 2022)
?
|
病毒產品 |
pAAV-EF1a-DIO-EYFP-WPRE |
|
實驗動物 |
?Ash1lfl/fl小鼠 |
|
注射方式 |
腦定位注射 |
|
感染部位 |
小鼠右側腦AUD區 |
|
注射體積 |
300nL |
?
DIO策略應用
Thy1是錐體神經元的特異性啟動子,在前腦、海馬、杏仁核、丘腦及視網膜等區域均有豐富的表達, Thy1-cre鼠配合rAAV載體可在以上腦區實現細胞特異性標記、光遺傳學和化學遺傳學操縱、在體鈣成像記錄,或者組織特異性的基因編輯。
?
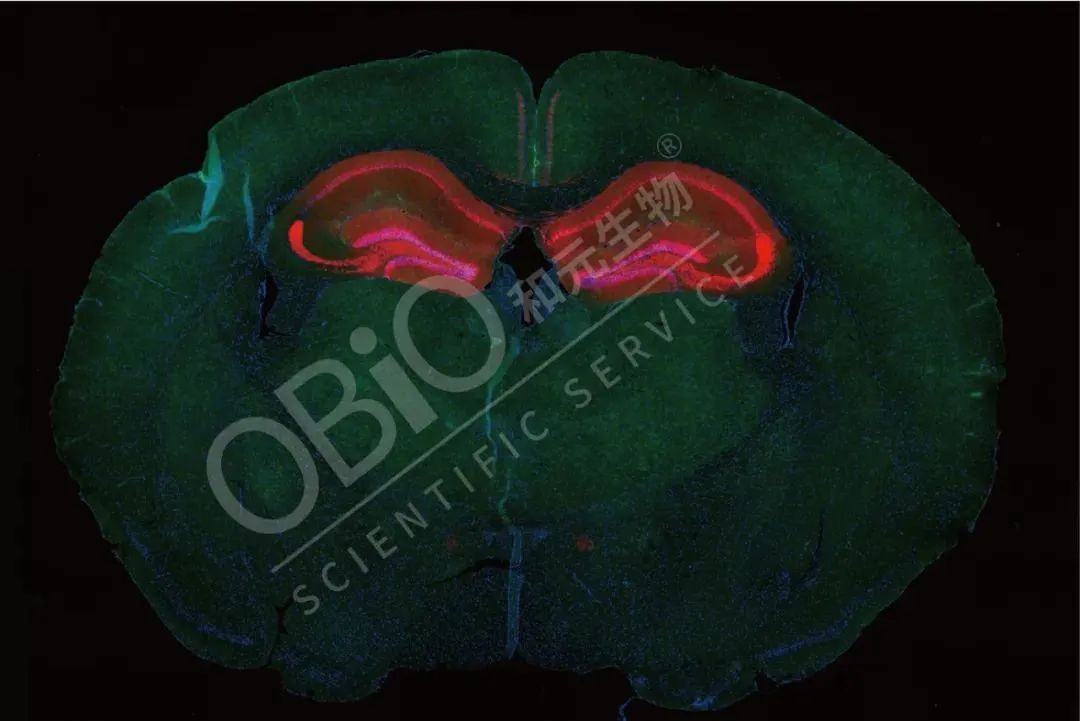
圖7 AAV-DIO注射Thy1-Cre鼠實現特異性標記
?
|
病毒產品 |
rAAV2/9-Ef1a-DIO-mCherry-WPRE |
|
實驗動物 |
Thy1-Cre小鼠 |
|
注射方式 |
腦定位注射 |
|
感染部位 |
小鼠雙側海馬 |
|
注射體積 |
200nL |
運用神經示蹤技術對大腦特定神經環路的結構和功能進行解析時,亦可以借助Cre重組酶系統和AAV血清型(比如rAAV2/9、rAAV2/retro、rAAV2/1)結合適用,達到特異性對神經環路標記和功能研究的目的。
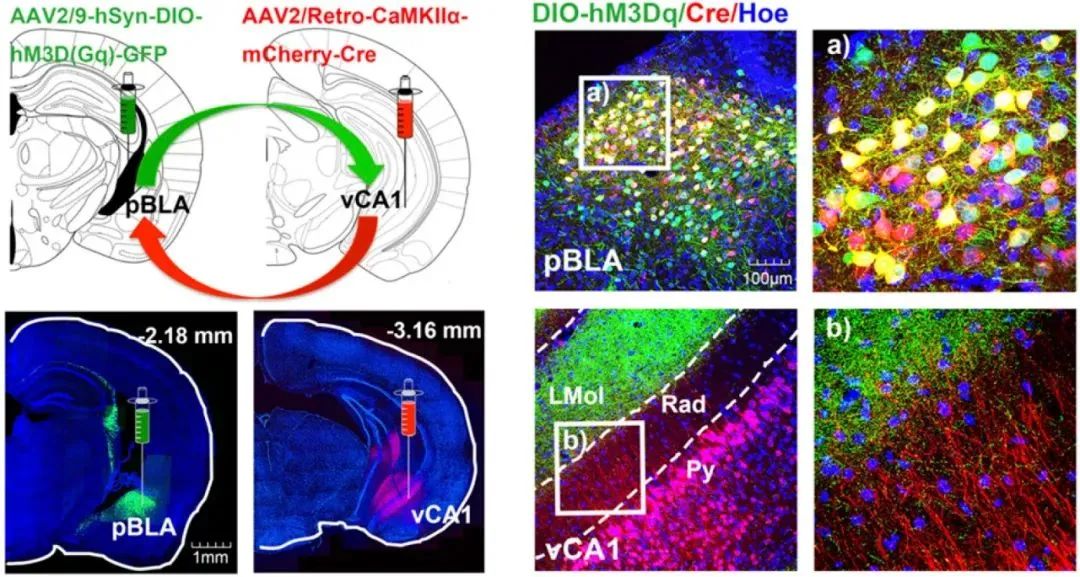
圖8 Cre-DIO系統結合病毒示蹤技術實現對特異環路標記及調控
(Ma Hui, et al., Proc Natl Acad Sci U S A., 2021)
|
病毒產品 |
rAAV2/Retro-CaMKIIα-mCherry-Cre |
|
實驗動物 |
小鼠 |
|
注射方式 |
腦定位注射 |
|
感染部位 |
小鼠vCA1和pBLA |
|
注射體積 |
200-300nL |
?
借助心肌細胞特異性啟動子Tnnt2驅動cre酶的表達,配合MADM-ML-11TG/GT鼠追蹤心肌細胞細胞分裂的狀態。(點擊閱讀:【Cell Stem Cell】雞尾酒療法促進心肌細胞增殖和心臟再生)。
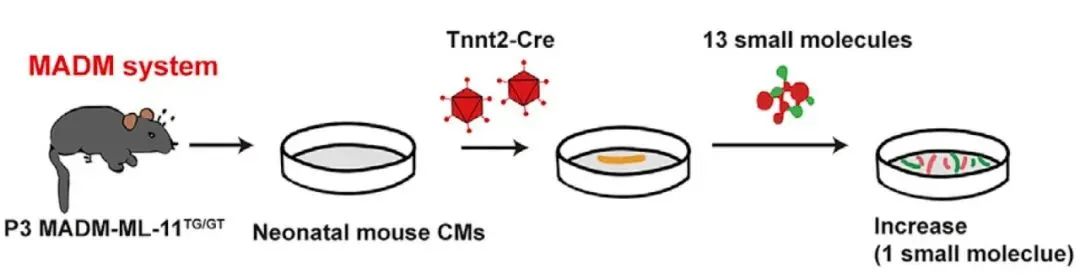
圖9 MADM系統應用
(Du Jianyong, et al., Cell Stem Cell., 2022)
|
病毒產品 |
pAdeno-Tnnt2-Cre |
|
實驗動物 |
P3 MADM-ML-11TG/GT鼠 |
|
感染部位 |
原代心肌細胞感染 |
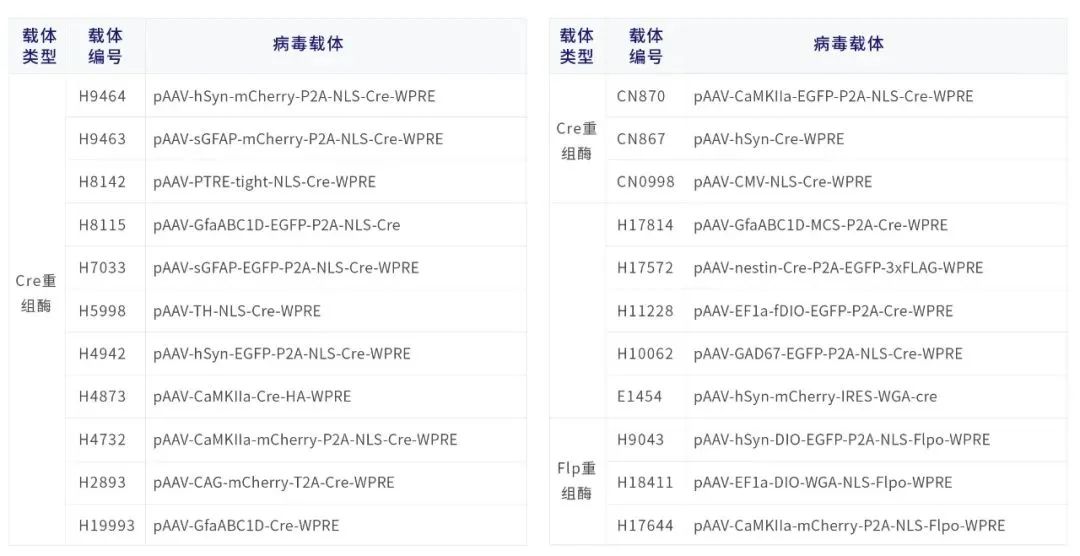
Cre病毒列表(部分)
改造的Cre/loxP系統
經過現代基因工程方法對Cre和loxP元件的改造,Cre/loxP系統實現了更加豐富的條件性重組策略。
1、對Cre元件的改造:對Cre元件的改造提高了Cre重組酶的活性,并且實現了藥物可誘導性。例如,通過在Cre元件上引入真核細胞核定位序列NLS,Cre能在低表達豐度下實現重組,這對于一些低豐度的啟動子很重要。另外,在Cre元件的C端接上一段改造過的配體結合結構域LBD,新的融合蛋白Cre-LBD將定位在胞漿內,當人工合成的激素分子結合到Cre-LBD受體后,蛋白構象改變并進入核內,介導基因重組,目前使用最多的是tamoxifen誘導的CreERT2突變體,它的LBD來自于雌激素受體ER分子,當有tamoxifen的時候,Cre才能介導基因重組,這樣通過控制tamoxifen的注射時間,可以實現對基因重組時間特異性的調控。
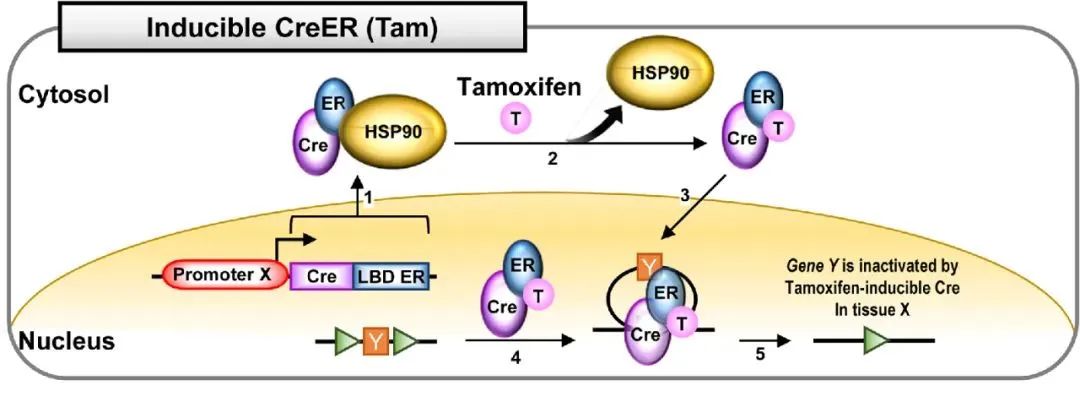
圖10 tamoxifen誘導的Cre-ER系統
(Hyeonhui Kim, et al., Lab Anim Res., 2018)
2、遠紅外光誘導的split Cre-loxP體系:化學誘導的Cre-loxP具有細胞毒性、泄漏、脫靶等劣勢,華東師范大學葉海峰研究員團隊基于split-Cre重組系統和遠紅外光(FRL)可誘導光遺傳系統建立了遠紅外光誘導的split Cre-loxP體系(FISC,far-red light-induced split Cre-loxP system)。在該體系中,Cre酶被分為兩個片段,N端Cre融合到一個Coh2結構(CreN-Coh2);C端Cre融合到一個DocS結構(DocS-CreC);FRL光照時,Coh2和DocS在強親和力的作用下結合,從而Cre酶也被重新組合,行使功能。該體系實現了條件性非侵入的基因調控目的。(點擊閱讀:【Nat Commun.】華師大葉海峰團隊創新研究:非侵入性遠紅外光誘導的Split-Cre系統調控基因重組)。
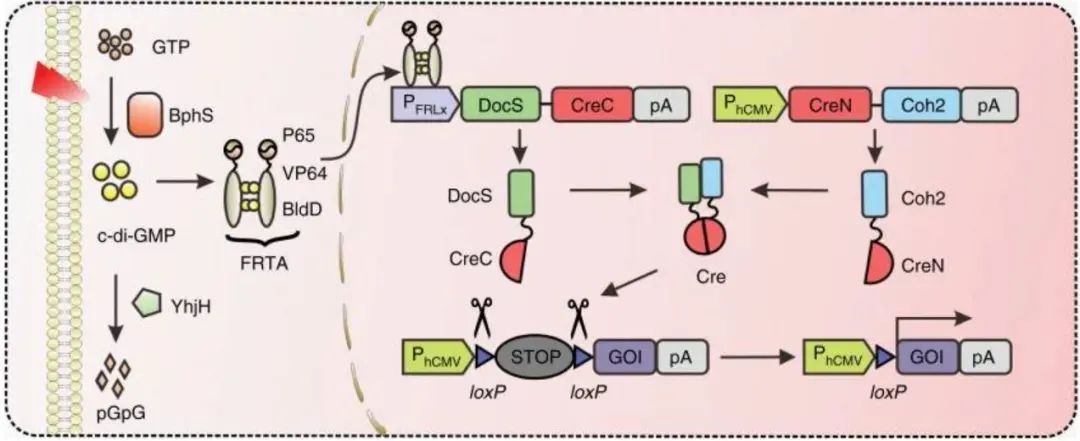
圖11 FISC體系
(Wu Jiali, et al., Nat Commun., 2020)
3、對loxP元件的改造:loxP元件也有一些突變體,間隔區和回文序列都可以進行突變,突變后的序列依然能被Cre識別和重組,但是突變的loxP序列必須和同樣突變的loxP序列匹配介導基因重組,而不能和未突變的loxP序列匹配,這樣將不同的loxP序列組合用于控制多個基因(如上文所述的lox2272位點),在同一Cre重組酶的作用下,可以實現多序列的基因重組,產生非常多元的重組結果。例如彩虹腦(Brainbow)技術絢麗多彩的熒光標記效果正式基于對loxP序列的改造實現。
?
其他重組酶系統
除Cre-LoxP系統外,類似的還有與Cre-LoxP系統無交叉影響的vCre-vLoxP、sCre-sLoxP系統,及Flp-FRT/F5系統和Dre-Rox1/Rox2系統,對應的表達方案分別為fDIO和dDIO系統。多套重組酶體系的存在為研究中設計多個限制條件提供了便利,靈活應用Cre、Flp、Dre重組酶系統將有助于更深入課題的開展(了解更多重組酶內容,點擊閱讀:【知識分享】一半海水、一半火焰,分子表達操控中高頻出現的Cre、Flp、Dre等重組酶系統如何工作)。
?
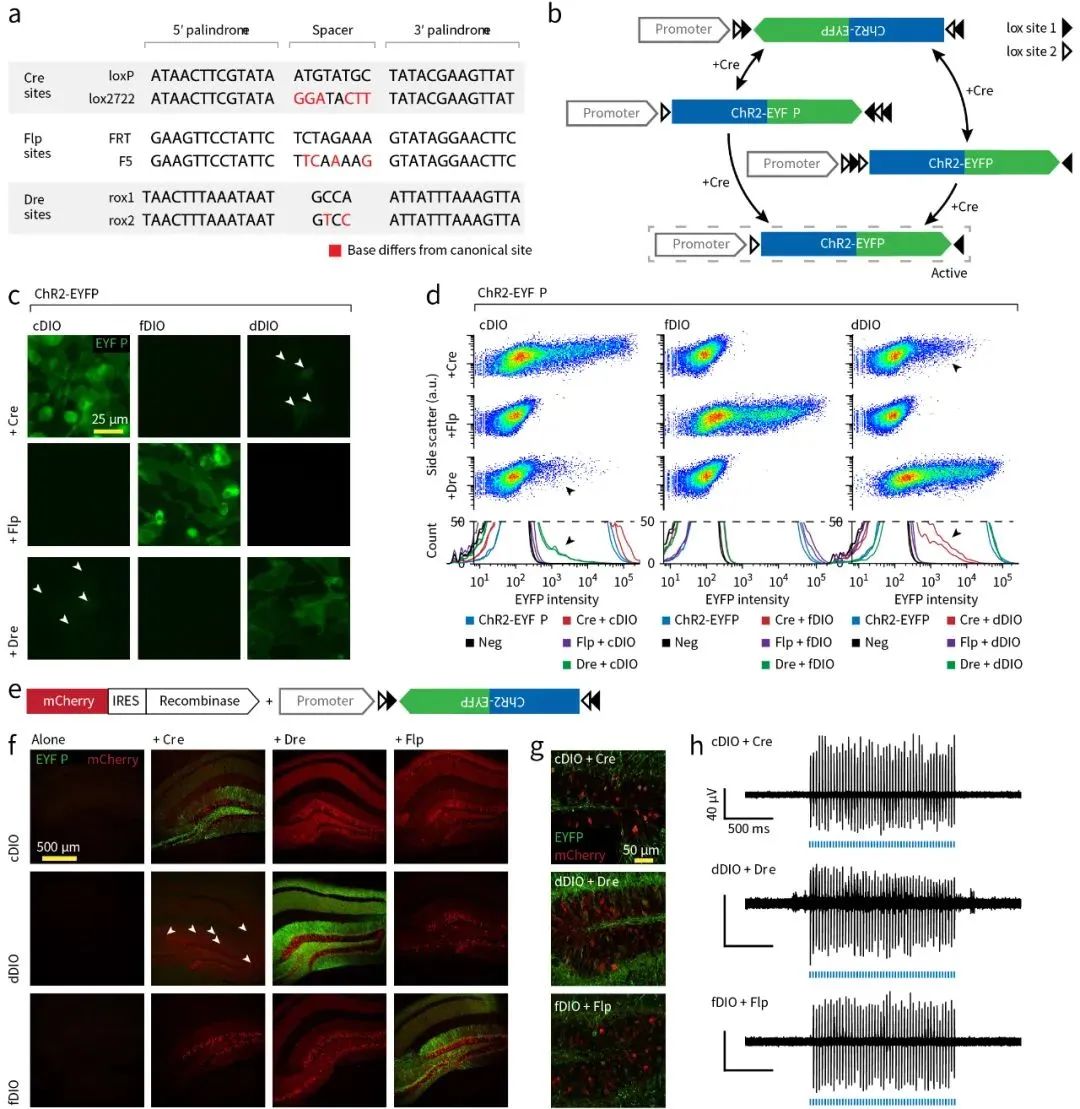
?
圖12 Cre\Flp\Dre系統比較
(Lief E Fenno, et al., Nat Methods., 2014)
參考文獻
[1] Front Genet. 2016 Feb 19;7:19. doi: 10.3389/fgene.2016.00019.
[2] Sci Adv. 2019 Feb 20;5(2):eaat3210. doi: 10.1126/sciadv.aat3210.
[3] Nat Methods. 2014 Jul;11(7):763-72. doi: 10.1038/nmeth.2996.
[4] J Biol Chem. 2020 Jan 17;295(3):690-700. doi: 10.1074/jbc.RA119.011349.
[5] J Neurosci. 2008 Jul 9; 28(28): 7025–7030.doi: 10.1523/JNEUROSCI.1954-08.2008
[6] Lab Anim Res. 2018 Dec;34(4):147-159. doi: 10.5625/lar.2018.34.4.147.
[7] Nucleic Acids Res. 2004 Nov 18;32(20):6086-95. doi: 10.1093/nar/gkh941.
[8] Dis Model Mech. Sep-Oct 2009;2(9-10):508-15. doi: 10.1242/dmm.003087.
[9] Nucleic Acids Res. 2011 Apr;39(8):e49. doi: 10.1093/nar/gkq1280.
[10]Cell. 2005 May 6;121(3):479-92.doi: 10.1016/j.cell.2005.02.012.
[11] Nat Commun. 2020; 11: 3708. doi: 10.1038/s41467-020-17530-9

