文章信息
論文標題:Identification of a Selective Polymerase Enables Detection of N6-methyladenosine in RNA.
刊登日期:2013年12月
發表雜志:Journal of the American Chemistry Society(美國化學協會雜志)
影響因子:14.375
研究機構:斯坦福大學化學系Kool課題組

對于想要具體對某一個m6A甲基化位置進行精確定位的老師,目前常用的方法是m6A-CLIP-seq從全基因組層面進行鑒定。另外,對于m6A甲基化程度較高的基因,絕大部分課題組采用MeRIP-seq的方法先初步確定基因的某個區域,然后再采用MeRIP-qPCR的方法進行后期驗證。這里我們將要介紹一種低通量驗證具體m6A甲基化位點的方法。來自斯坦福大學化學系的Kool教授團隊,在2013年成功從極端嗜熱菌Thermus thermophilus中鑒定出一種逆轉錄聚合酶。這種特殊的聚合酶,針對未發生m6A修飾互補配對的T堿基具有高達18倍的選擇性。換句話說,這種聚合酶一旦發現具有m6A修飾的腺嘌呤A后,逆轉錄擴增將會停止。Kool教授認為,這種酶通過分析電泳條帶可以對RNA中m6A具體修飾位置進行定位及定量分析。此外這種酶與非特異選擇性聚合酶(nonselective polymerase)在性能上進行了比較,分析兩者在驗證高豐度m6A修飾RNA的區別。最后Kool教授在28s rRNA上的第4190位精確定位到了m6A修飾。
鑒定m6A修飾的功能是一項艱巨的任務。早期不少實驗室采用酶消化和同位素放射性標記的方法來鑒定m6A所在的RAC motif。這些研究還表明,在任何位點的m6A甲基化水平都是不太完整的,通常結果也只能說明RNA某個位點m6A修飾水平在22-99%之間,無法做到100%。
高通量測序的技術進步與抗體特異性富集等方法相結合后,不少課題組對傳統的RIP-seq進行了針對m6A課題研究的改良和升級——MeRIP-seq。這種方法的優勢在于可以幾乎從全基因組層面鑒定所有的mRNA、lncRNA和circRNA等RNA上的m6A修飾情況。不過這種方法通常只能對高甲基化區域進行鑒定,而不能具體到某個m6A位點。
在此基礎上,miCLIP-seq以及CLIP的升級版enhanced CLIP由此誕生。前者會涉及到同位素標記,而后者在犧牲部分精度情況上能夠大大提升實驗的通量且無需同位素標記。
然而,上面提到的所有方法都需要在酶消化的幫助下才能進行。若沒有酶的消化,會出現腺嘌呤上的堿基修飾情況仍然精度不夠等缺點。所以判斷RNA在哪個堿基處斷裂(RNA長度不同導致后期電泳長度不同)無疑是一種幾乎不需要借助酶消化就能達到目的的方法,這時候,咱們提到的聚合酶就要登場了。作者推測極有可能存在一種只對m6A具有顯著特異選擇性的聚合酶,并且這種酶可能被用于位點特異性修飾檢測。
作者終于在一大堆細菌的candidate聚合酶中,找到了這種特殊的聚合酶。這種特異性逆轉錄聚合酶取自一種極端嗜熱菌Thermus thermophilus中。聚合酶因其對堿基的空間結構極其敏感,所以是一種新型的堿基修飾定位驗證機制。
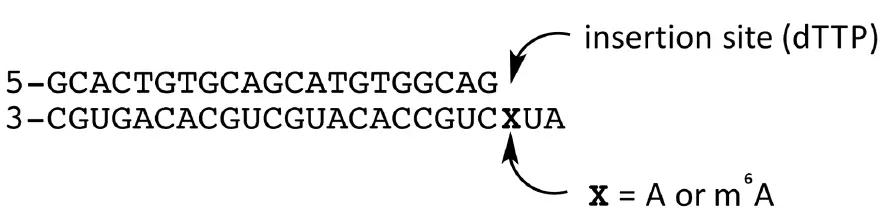
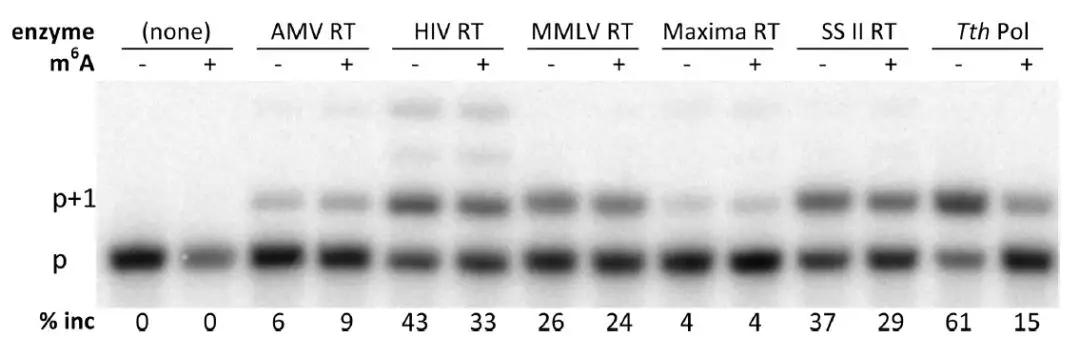
那么他們究竟是怎么做的呢?首先作者對大量具有逆轉錄酶活性的candidate進行第一步篩選。RNA模板設置有兩組,一組帶m6A修飾一組不帶m6A修飾,以此作為底物模板來監測這些酶是否對被同位素標記的三磷酸胸苷(dTTP)DNA引物有延伸能力。在諸多的逆轉錄酶中,嗜熱菌Tth在所有參與測試的酶中表現出較強的選擇性,并在Mn2+存在的情況下充當逆轉錄酶的角色。
由于Tth酶有在某個特定條件下對一個特定的RNA模板顯示出極強的特異選擇性,所以作者進一步測試了不同溫度、反應時間長短以及buffer成分是否可能會增強其特異選擇性。已知Mn2+能夠降低酶選擇性,然而作者卻發現Mn2+對于Tth酶的逆轉錄聚合酶活性至關重要。
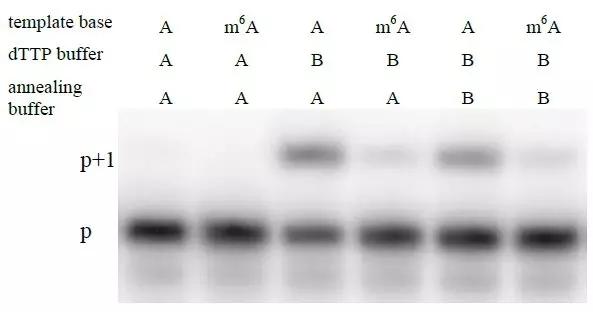
下一步,作者想要驗證這種酶是否對其他序列具有延伸性。作者將小鼠的eef2基因的3’ UTR以及其他基因序列作為模板底物,這個模板分為帶m6A修飾和不帶m6A修飾的兩份,并進行酶活性動力學驗證。
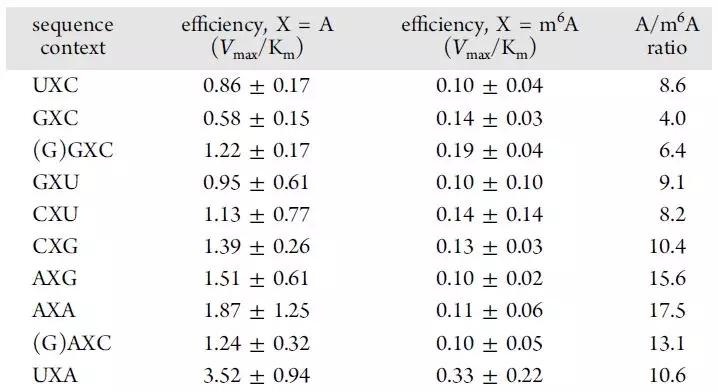
針對有m6A修飾的序列模板,在不含m6A修飾的對照組中Tth酶具有4-18倍的效率。整體上,不同背景下選擇性差異可以忽略不計。例如T互補的A堿基在未發生修飾情況下,UAA序列相比其他序列具有更高的效率。此外,5’ G和3’ C都出現酶效率降低的情況。最值得注意的是,針對GAC motif,Tth聚合酶的活性提高了4-6.4倍。因此,這些現象均表明酶的選擇性識別m6A修飾的motif是一個十分普遍的現象。
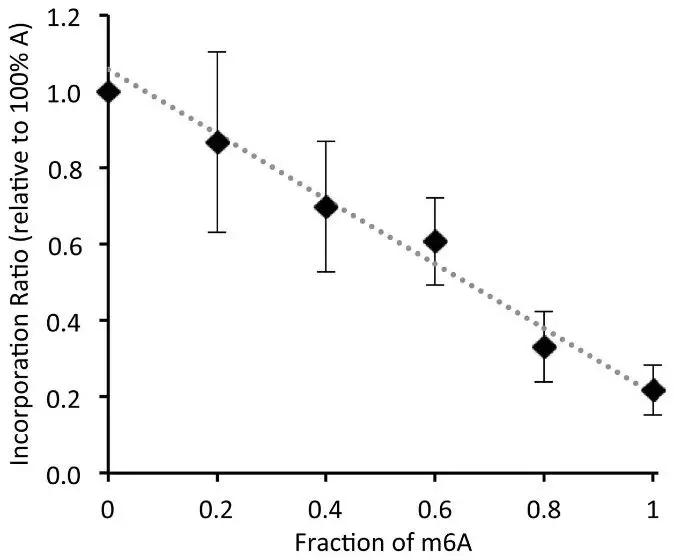
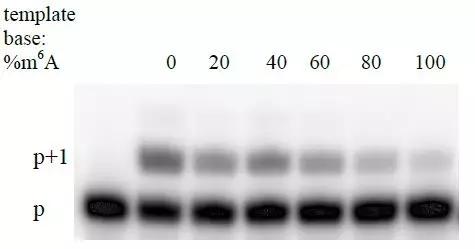
接下來作者要驗證Tth酶可否對具體某個A堿基的m6A修飾程度進行定量。作者混入了已知m6A比例的RNA作為模板,在一個固定的時間點對dTTP消耗量進行測量。在已知的這份RNA模板中引物延伸的比例呈現出線性關系,這表明Tth酶可用于對具體的某個m6A甲基化位點進行定量。
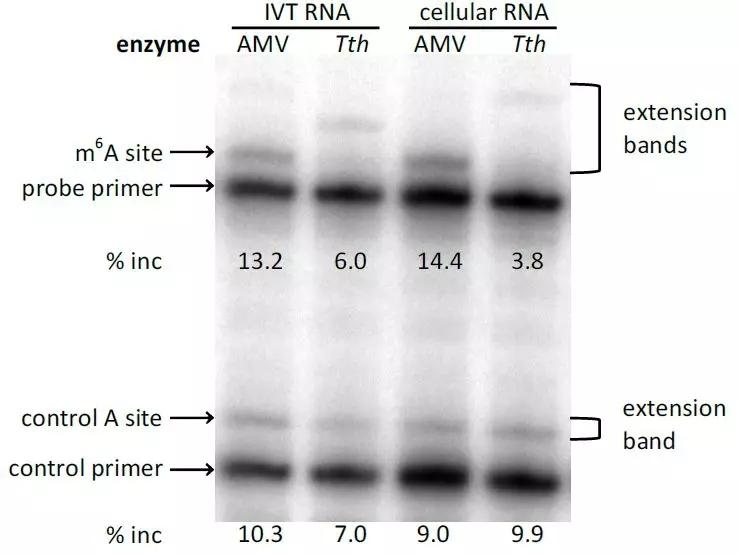
作者進一步驗證Tth酶可否用于檢測從哺乳動物細胞提取RNA中的m6A甲基化水平。由于RNA本身的濃度及其二級結構可能影響引物的延伸效率,作者引入了兩種控制實驗反應的方法。其中第一個策略是使用兩種可以同時反應的引物(一個緊挨著被探測的甲基化位點,一個與已知的甲基化狀態的堿基相鄰)。這兩種引物能夠對樣本中已知的RNA量進行測量。第二個策略使用的是非特異性聚合酶(即AMV RT逆轉錄酶)與同一種引物進行反應。之所以使用AMV RT逆轉錄酶是因為,在不同比例的RNA濃度下,AMV RT酶對Tth酶呈現線性相關。在感興趣的堿基位點插入T后可用于檢測非特異性酶AMV RT與Tth酶之間的區別。
已知人的核糖體RNA(rRNA)上的A堿基含有m6A修飾,包括18s rRNA的1832位,28s rRNA上的4189和4190位。針對鄰近未發生堿基修飾進行酶消化后的比例,上面所述的幾個rRNA的位點幾乎存在100%的m6A修飾。作者針對這三個發生m6A修飾的位點設計了引物。從293細胞中提取total RNA,并對引物覆蓋到的RNA位點進行標記。
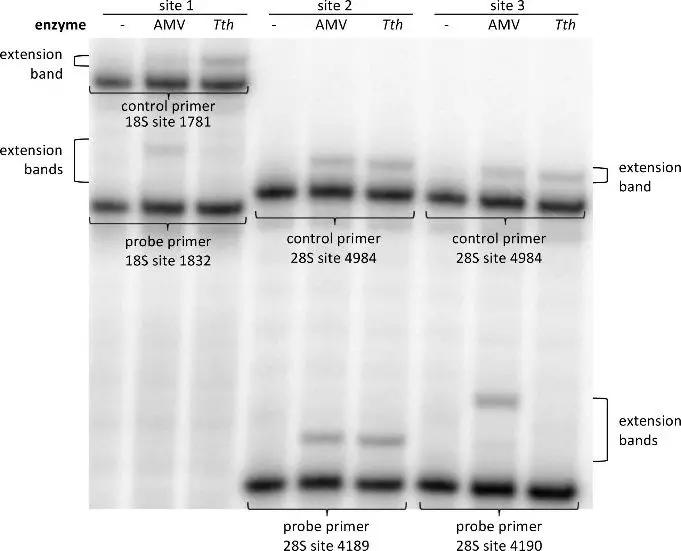
如上圖顯示,與對照組相比Tth組rRNA中1832和4190這兩個位點對T的親和度降低20%,并且針對這個作者使用人工合成的RNA進行了定量驗證。與之相反的是,作者觀察到相對于AMV RT逆轉錄酶,Tth酶對rRNA中4189位點的親和度比對照組高出120%。因此作者認為未發生甲基化的位點就是非常明確地位于28s rRNA的4190而不是4189附近。
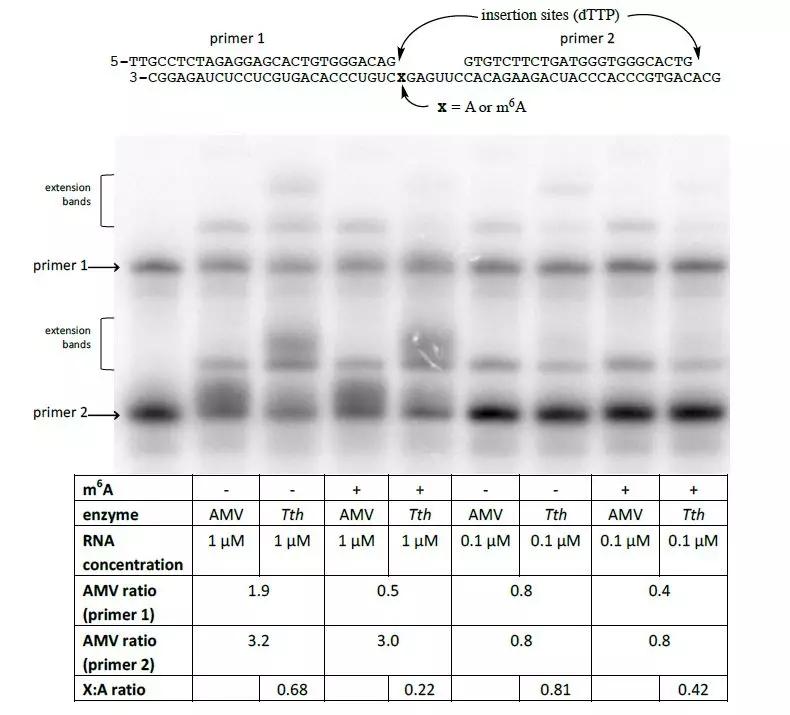
最后,作者試圖在哺乳動物細胞中提取的mRNA中驗證m6A修飾的具體位點。下一步作者選擇bRPL基因3’ UTR序列上已知的m6A位點。已知bRPL的3’ UTR上m6A修飾水平在20%左右,作者在293細胞中過表達bRPL的3’ UTR序列。接下來設計兩對特異性引物,檢測已知的修飾位點以及腺嘌呤附近的堿基。
當RNA上含有m6A修飾的堿基位點時,親和性顯著低于不帶任何m6A修飾的堿基。作為額外對照組,作者在體外表達了一個不帶m6A修飾的bRPL,檢測引物對其的親和度。前期濃度較高的時候實際上,無論是細胞中提取的total RNA還是體外轉錄的RNA基本沒有差異。然而在酶的濃度比較低時,m6A就發生了比較大的差異。這就說明293細胞中確實存在m6A修飾。
