基因編輯是一項革命性的技術,但其脫靶效應一直是科學家對CRISPR-Cas臨床應用的擔憂。基因編輯可能導致在脫靶位點發生小片段的插入缺失,還有可能在在靶位點發生意外的大片段刪除或染色體倒位、易位。染色體重排異位非傳統意義的脫靶,但是可造成染色體劇烈改變,引起嚴重的毒副反應。美國FDA對基因治療產品的指導原則《Human Gene Therapy Products IncorporatingHuman Genome Editing》里明確指出,臨床基因治療需要評估脫靶導致的染色體易位。

?圖1: FDA關于染色體重排的指導原則
在此,舒桐科技小編向大家介紹一種新的染色體重排檢測技術——Primer extension-mediated sequencing (PEM-seq) ,它可以全面、精確量化基因編輯的雙鏈斷裂(DSB)后DNA的修復結果。
PEM-seq的實驗設計非常巧妙,通過在DSB上游設計引物(稱為誘餌primer),捕獲下游與DSB位點DNA修復后連接的序列(稱為獵物序列)。與其他脫靶分析技術相比,PEM-seq的優勢在于它可以檢測到在靶位點的小片段和大片段的插入缺失(indel),以及由脫靶導致染色體易位。PEM-seq的詳細的實驗步驟如下圖2。
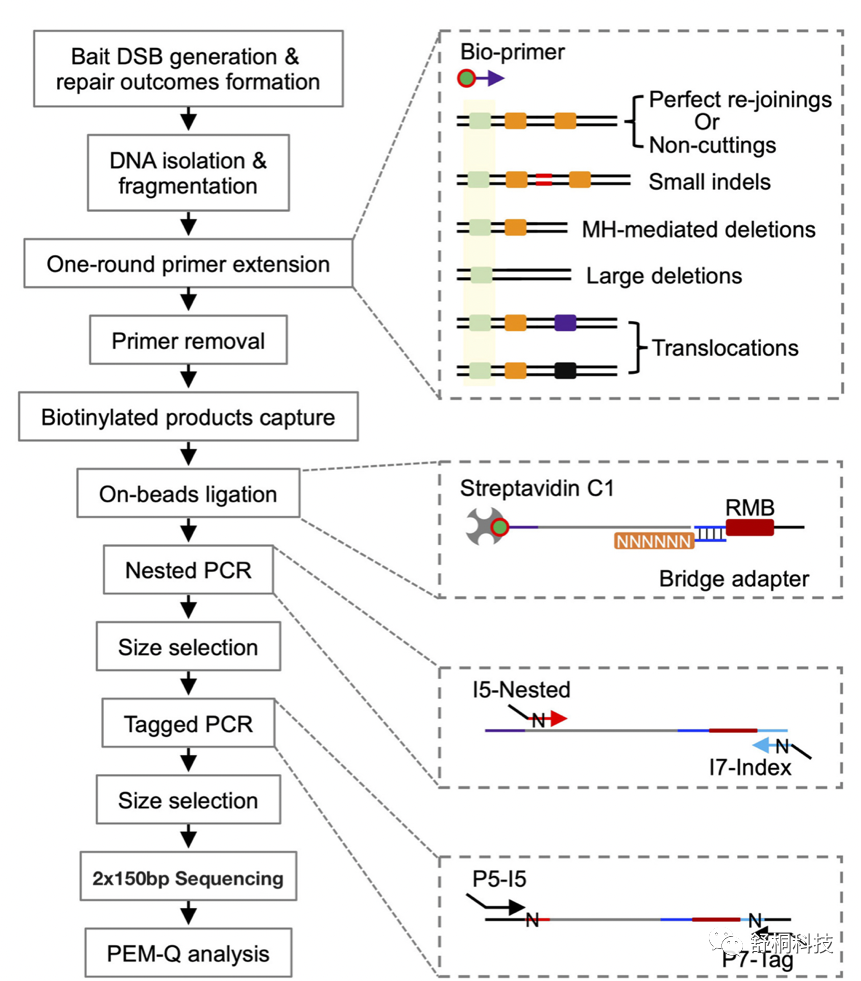
圖2 PEM-seq實驗原理。
首先,PEM-seq可以得到在靶indel的編輯效率,并且與公認的擴增子編輯分析軟件CRISPResso結果相關性達到0.99,如下圖3。
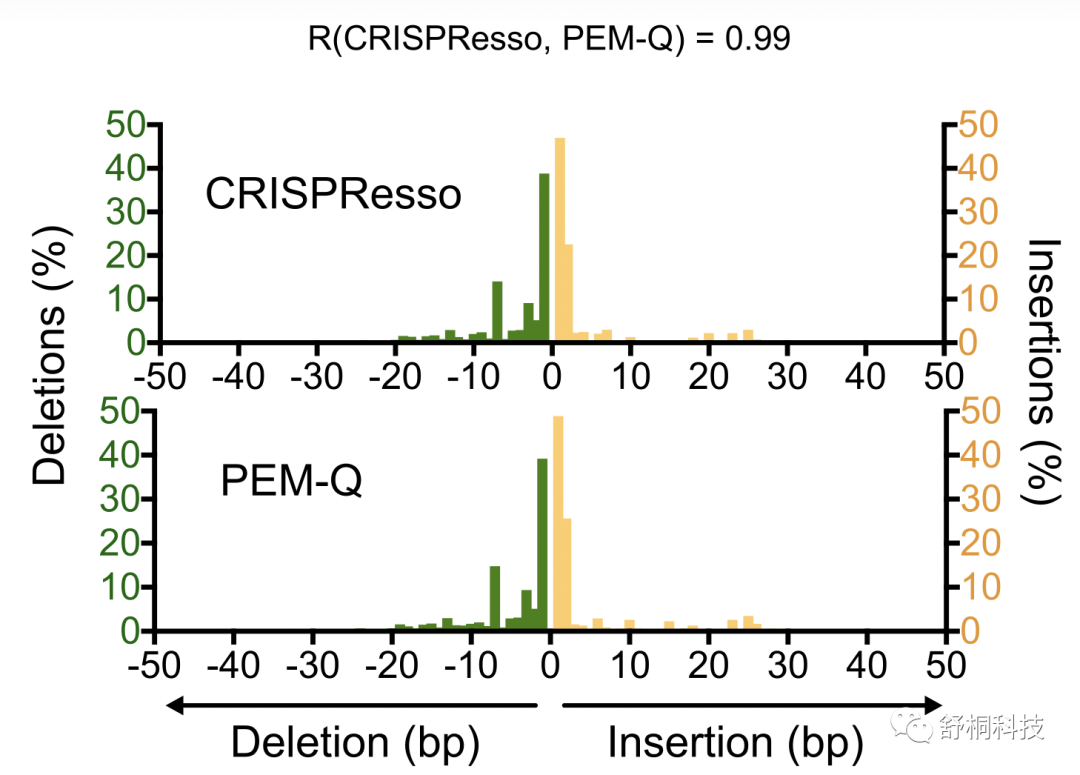
圖3 在靶位點小片段插入缺失分布和與CRISPResso對比。來源:Nucleic acids research?49.15 (2021): 8732-8742.
?其次,PEM-seq通過捕獲還可以得到大片段的插入(>=20bp)和刪除(>=100bp)信息,這是常規擴增子無法得到的。如下圖4、5。

圖4 在靶位點大片段插入分布。來源:Nucleic acids research?49.15 (2021): 8732-8742.

圖5 在靶位點大片段刪除分布。來源:Nucleic acids research?49.15 (2021): 8732-8742.
更進一步,如果捕獲到的獵物序列距離DSB位點>500kb,那么這些位點則是易位產生的,誘餌Primer捕獲到的基因組易位位點通過下面的Circos圖展示。通過篩選易位位點與sgRNA同源序列,即可得到脫靶位點。如圖6,SpCas9,在基因組易位位點中共發現了7個脫靶導致的易位,而AsCas12a沒有檢測到脫靶導致的易位。
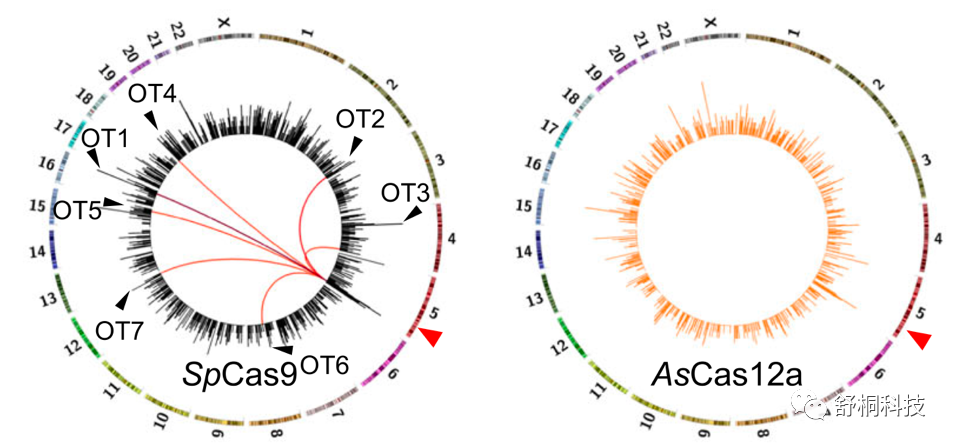
圖6 誘餌Primer捕獲到的易位位點(外圈),以及脫靶易位位點(內圈為在靶和脫靶位點連線)。來源:Nature Communications?13.1 (2022): 5623.
?目前,PEM-seq已應用于描繪Cas12家族的編輯產物的特征,該技術實現了對基因編輯的全面、高精度定量的安全性評估,在分析基因編輯的大片段插入、刪除、易位展現了獨特的優勢。因此,PEM-seq有望成為基因編輯領域的標準化脫靶分析技術,并為新型基因編輯工具臨床應用提供前瞻性指導。
PEM-seq科研服務
舒桐科技跟蹤最新前沿進展,配合資深研發團隊現已推出PEM-seq實驗和分析服務,服務詳情可見舒桐科技官網。
關于舒桐
珠海舒桐醫療科技有限公司深耕基因編輯技術10余年,是一家以基因編輯為核心技術的生物醫藥服務企業,可承接脫靶檢測、細胞株定制、病毒包裝等CRO及CDMO業務。目前,舒桐科技擁有3000㎡的GMP研發實驗室及生產車間;擁有博士后科研工作站及澳門理工大學教學科研實習基地。研發團隊熟練掌握各類基因編輯技術,包括基因編輯、堿基編輯及先導編輯等,成功完成如基因敲除、點突變、大片段插入、過表達等細胞系構建項目;開發了多種具有自主知識產權的脫靶檢測技術,建立了高通量的sgRNA篩選平臺;建立了工廠化新型CRISPR挖掘體系,已開發出多種切割效率高且低脫靶的新型CRISPR。公司目前已申請基因編輯、脫靶檢測、新型CRISPR及藥物遞送等**70+項,授權50+項。同時,公司具備先進的CGT原料規模化生產能力,產品質量達到**水平,可為科研機構及生物醫藥企業提供安全高效的一站式服務和原料供應。舒桐科技堅持以“創新、敬業、融合、開放”為核心價值觀,致力于利用基因治療技術造福人類健康,讓生命更美好。
如需獲得更多信息,請咨詢我們:
電話:
400-6309596
15022705442
企業官網:
http://www.generulor.com
產品訂購/技術支持:
service@generulor.com
參考文獻:
1.Liu, Mengzhu, et al. "Global detection of DNA repair outcomes induced by CRISPR–Cas9."?Nucleic acids research?49.15 (2021): 8732-8742.
2. Xin, Changchang, et al. "Comprehensive assessment of miniature CRISPR-Cas12f nucleases for gene disruption."?Nature Communications?13.1 (2022): 5623.
