細菌基因組de novo測序
產品名稱: 細菌基因組de novo測序
英文名稱: Bacterial genome de novo sequencing
產品編號:
產品價格: 詢價
產品產地: null
品牌商標: null
更新時間: null
使用范圍: null
- 聯系人 :
- 地址 : 上海市浦東新區國際醫學園區康新公路3399弄3號樓
- 郵編 : 201203
- 所在區域 : 上海
- 電話 : 159****9102 點擊查看
- 傳真 : 點擊查看
- 郵箱 : marketing@majorbio.com
產品介紹
隨著高通量測序技術和生物信息學的快速發展,細菌全基因組denovo測序已然成為一種探究細菌生物學問題的高性價比方法。通過全基因組測序,可以獲知待測菌株的基因及相關調控信息,為研究該菌株特有的生物學特征(致病機制,共生機制,獨有的代謝機制)提供分子基礎;通過比較基因組分析,可以研究種內及種間的相互進化關系,為探究疾病的傳播機制提供理論指導。
目前,細菌基因組測序已經廣泛應用于細菌流行病學、疫苗開發、微生物進化等多個領域。此外,細菌全基因組de?novo測序也能為該物種后續基因組數據的挖掘以及重要基因的功能分析構建一個全面的研究平臺。
美吉優勢
擁有標準化操作實驗室和高通量測序技術平臺,實驗周期短,保證高質量的數據;
擁有多種測序平臺和強大的計算機資源,提供最佳的測序解決方案和快速的分析平臺;
擁有專業的分析團隊,全面的分析內容,提供全面和高質量的基因組解析結果;
產品類型
?
?? 細菌基因組框架圖
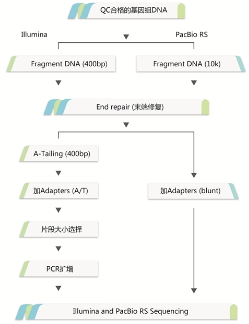
????? 構建Illumina?PE(~400bp)文庫,通過Illumina(Miseq/Hiseq)平臺測序并進行de?novo基因組組裝,初步獲得基因組序列和功能注釋信息。
? 細菌基因組完成圖
??? 構建PacBio?RS?II(~10K)文庫,基于多種測序平臺(ABI3730xl,Illumina?Miseq/Hiseq,Pacbio?RS?II)獲得基因組完成圖并進行深度生物信息挖掘。
實驗流程
?
? 框架圖分析流程:???????????????????????????完成圖實驗流程: ? 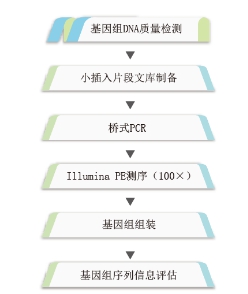
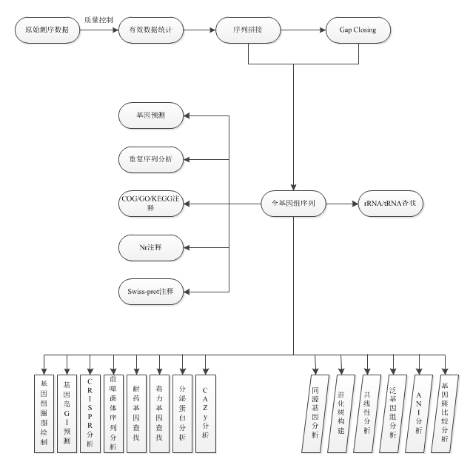
生信分析
?
生信分析內容:
?
|
高級分析I(序列分析) |
高級分析II(比較基因組) | |
|
原始數據統計與處理 |
基因圈圖繪制 |
同源基因分析 |
|
基因組拼接 |
基因島(GIs)預測 |
基因組進化樹構建 |
|
?基因預測 |
CRISPR分析 |
共線性分析 |
|
重復序列分析 |
前噬菌體序列分析 |
泛基因組分析 |
|
非編碼RNA預測 |
耐藥和毒力基因注釋 |
ANI分析 |
|
Nr/Swiss-prot注釋 |
分泌蛋白分析 |
基因簇比較作圖分析 |
|
COG/GO/KEGG注釋 |
碳水化合物相關酶數據庫(CAZy)注釋 |
技術指標
????框架圖承諾指標:
????? 基因組覆蓋度大于95%,基因區覆蓋度98%以上,單堿基錯誤率低于十萬分之一
?? 完成圖承諾指標:
????? 0?GAP,1?contig。
送樣要求:
???? 1)樣品類型:?DNA/菌體
???? 2)樣品需求量(單次):菌體樣本:≥5g;?基因組DNA:≥2μg(框架圖),基因組DNA:≥10μg(完成圖)
???? 3)樣品濃度:≥20ng/μL
?? ??4)樣品純度:OD260/280=1.8-2.0并確保DNA無降解,無污染。
??? 5)樣品保存期間切忌反復凍融,送樣時請使用冰袋或干冰運輸。
案例解讀
案例一:基因組分析揭示了Bacillus?cereus?Group中各菌株的分類地位[1]
本文利用224株芽孢桿菌基因組序列,通過GBDP/dDDH、16S?rRNA以及特殊基因MLSA等方法進行芽孢桿菌分類地位研究。解決幾個問題:1)比較這些方法的分類結果;2)重新構建蠟樣芽孢桿菌的系統進化樹;3)探索芽孢桿菌的遺傳多樣性;4)獲得更滿意的分類結果。
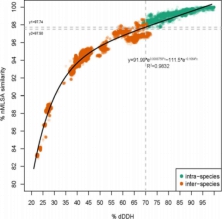
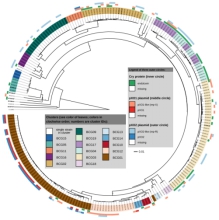
圖1?dDDH值與nMLSA相似性的相關性分析????圖2?pycA基因構建系統進化樹
案例二:213株乳桿菌的比較基因組學分析[2]
本文對213株乳桿菌屬模式菌株,采用二代Illumina?HiSeq?2000高通量測序平臺繪制其基因組精細圖譜,結合比較基因組和功能基因組分析方法,解析與菌株分化相伴隨的功能基因進化歷程,拓展其生物應用潛力。
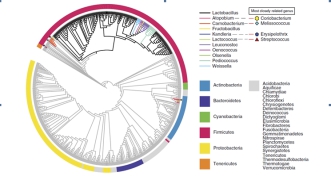

????????????????????????????????? 圖1基于16個持家基因的最大似然法系統進化樹???????????????? ?圖2基于73個核心基因的系統進化樹
參考文獻
[1]?Liu?Y,?Lai?Q,?G?ker?M,?et?al.?Genomic?insights?into?the?taxonomic?status?of?the?Bacillus?cereus?group.?Scientific?Reports,?2015,?5:?14082-14082.
[2]?Sun?Z,?Harris?H?M?B,?McCann?A,?et?al.?Expanding?the?biotechnology?potential?of?lactobacilli?through?comparative?genomics?of?213?strains?and?associated?genera.?Nature?Communications,?2015,?6:?8322.
常見問題
(1)細菌基因組組裝效果的指標有那些?在一個什么樣的范圍算是比較正常的?
我們進行多個k-mer參數的拼接后,會綜合考慮scaffold?N50長度值、N%、scaffold數量、總堿基數等指標,一般要求N50較長,N%較低,scaffold數量相對較少,這樣拼接比較集中,不至于太零散;依據這些技術指標進行綜合評定,來選取最終的組裝結果;因為不同菌的基因組有差異,分析要達到的精細程度也不同,所以會根據客戶要求和基因組情況選取最佳結果,各個指標的范圍也不同。
?
(2)對于GC含量過高或過低以及重復序列較多的基因組,完成圖能否0Gap?
?? GC含量大于65%或者小于35%以及重復序列較多時,會對測序和組裝有一定影響,但目前Pacbio長讀長的測序技術能很好的克服這些問題,美吉生物有幾百例的細菌完成圖項目經驗,利用三代測序技術全部都可以達到0gap的結果。
